İçindekiler:
- Yazar John Day day@howwhatproduce.com.
- Public 2024-01-30 13:17.
- Son düzenleme 2025-01-23 15:13.
Yazan: Phuc Lam, Paul Yeung, Eric Reyes
Akciğer segmentasyonundaki hataların, bir hastalık bölgesinin tanımlanmasıyla ilgili yanlış bilgiler üreteceğini ve teşhis sürecini doğrudan etkileyebileceğini kabul etmek. Akciğer hastalıkları zorlu şekillere sahip olduğunda modern bilgisayar yardımı teknikleri doğru sonuçlar vermede başarısız oldu. Bu anormal şekillere plevral efüzyonlar, konsolidasyonlar vb. neden olabilir. Akciğer sınırlarının çevreleyen torasik dokudan izole edildiği akciğer segmentasyon tekniğini uygulayarak uygulamamız, tamamen özelleştirilebilir görünümler sağlamak için sınırları kullanıcının giriş eşikleriyle tanımlayabilir. akciğerlerin şekillerinden, Bu MatLab projesinin amacı, akciğerlerin röntgen görüntülerinin patolojik durumlarını tespit etmek için kullanıcı dostu etkileşimli bir akciğer segmentasyonu uygulaması oluşturmaktır. Amacımız, doktorlara ve radyologlara akciğer hastalıklarını teşhis etmede daha güvenilir bir yol sağlamak için anormal akciğerleri göstermenin ve tanımlamanın daha etkili bir yolunu yaratmaktır. MatLab'daki uygulama tasarımcısı aracını kullanan program, özellikle göğüs röntgeni ve bilgisayarlı tomografi (BT) taramalarıyla çalışmak üzere tasarlanmıştır, ancak MRI taramalarıyla da çalışacak şekilde test edilmiştir.
Aşağıdaki talimatlar gürültü filtreleme tekniğimizi (düşük geçişli Wiener filtresi) ve görüntü eşiğini (gri tonlamalı görüntünün yoğunluk histogramını kullanarak) ve bir morfolojik gradyan (bir görüntünün genişlemesi ve aşınması arasındaki fark) kullanarak içerir. bir ilgi alanı belirleyin. Talimat daha sonra tüm öğeleri grafik kullanıcı arayüzüne (GUI) nasıl entegre ettiğimizi açıklayacaktır.
Not:
1). Bu proje bir araştırma makalesinden esinlenmiştir: "CT'de Anormal Akciğerlerin Segmentasyonu ve Görüntü Analizi: Mevcut Yaklaşımlar, Zorluklar ve Gelecek Eğilimler". Hangisi burada bulunabilir
2). NIH: Clinical Center'dan X-ray görüntüleri kullanıyoruz. Bağlantı burada bulunabilir
3). Uygulama tasarımcısı yardımı burada bulunabilir
4). Kodu çalıştırmadan önce: Dir yolunu (34. satırda) dosya dizininize ve görüntü tipine (35. satır) (biz *-p.webp
Adım 1: Adım 1: Görüntüyü Yükleme
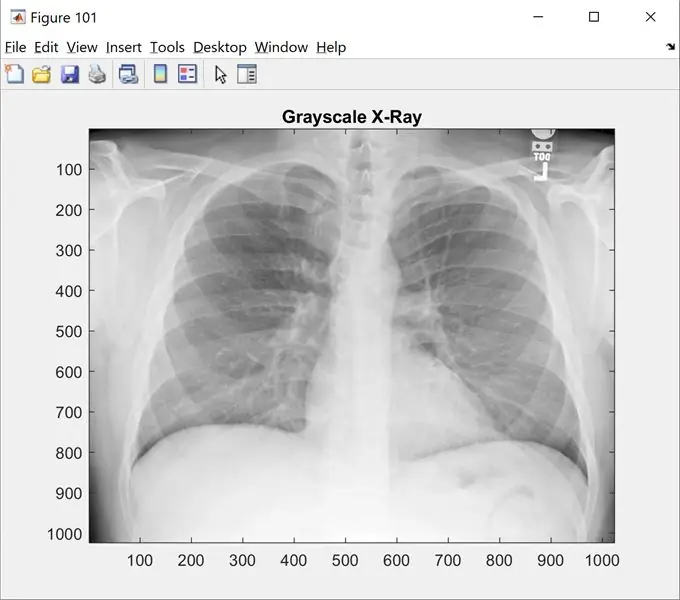
Bu adım size orijinal resmi gri tonlamalı olarak gösterecektir. 'name_of_picture.png' dosyasını resim adınızla değiştirin
açık; clc; hepsini kapat;
%% Resimler yükleniyor
raw_x_ray='name_of_picture.png';
I=imread(raw_x_ray);
şekil(101);
imshow(I);
renk haritası(gri);
title('Gri Tonlamalı Röntgen');
Adım 2: Adım 2: Gürültü Filtreleme ve Histogram
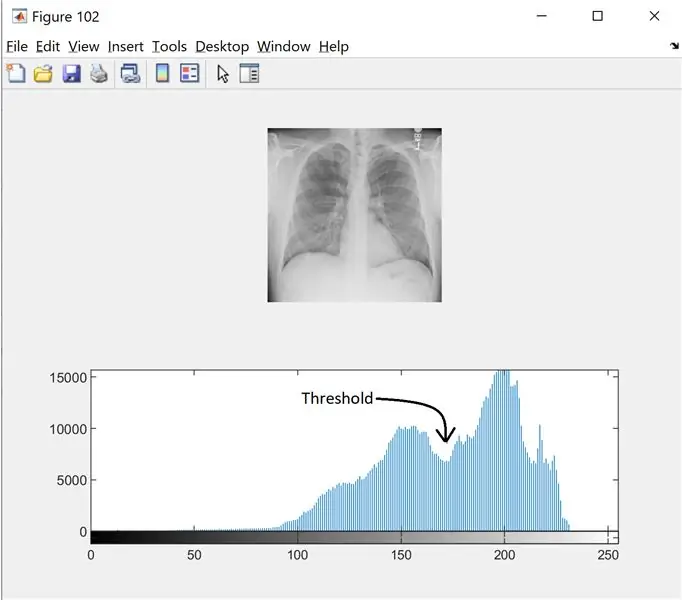
Gri tonlamalı görüntünün eşiğini bulmak için, farklı modlar olup olmadığını görmek için histograma bakarız. Daha fazlasını buradan okuyun
I=wiener2(I, [5 5]);
şekil(102);
subplot(2, 1, 1);
imshow(I);
alt grafik(2, 1, 2);
imhist(I, 256);
Adım 3: Adım 3: Eşiklerin Ayarlanması
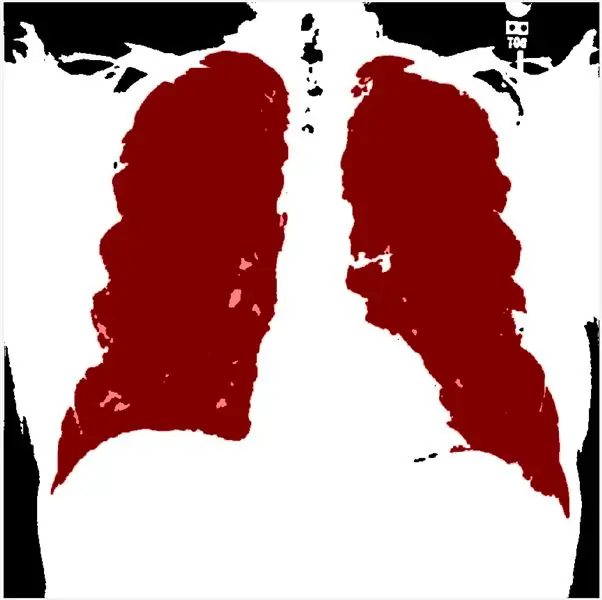
Bu adım, histograma göre eşiği ayarlamanızı sağlar. morfolojikGradient, ilgilenilen bölgeyi kırmızıyla vurgulayacaktır ve işlev görsel sınırları, akciğerin ana hatlarıyla belirtilen ve filtrelenmiş görüntüsünü kırmızıyla kaplar.
Regionprops kullanarak, katılık dizilerini kesinleştirebilir ve azalan şekilde sıralayabiliriz. Sonra gri slae görüntüsünü ikili hale getiriyorum ve ilgilenilen bölgeyi (ROI) vurgulamak için morfolojik gradyan yöntemini ve mLoren Shurasking'i uyguluyorum. Bir sonraki adım, görüntüyü ters çevirerek akciğer ROI'sinin siyah arka planda beyaz olmasını sağlamaktır. 2 maskeyi görüntülemek için showMaskAsOverlay işlevini kullanıyorum. Not: kod, Loren Shure'dan ilham almıştır, bağlantı.
Lasly, bwbwsınırlarını kullanarak ve filtre görüntüsünü ve sınırlarını maskeleyerek kırmızı anahat oluşturuyorum.
a_thresh = I >= 172; % bu eşiği ayarla
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
sahne = bölgeprops(a_thresh, 'tümü');
sortedSolidity = sort([props. Solidity], 'iniş');
SB = sıralanmışSolidity(1);
SB == %1 ise SB yalnızca katılığı kabul eder == 1 kemikleri filtreler
binaryImage = imbinarize(I); şekil(103);
imshow(binaryImage); renk haritası(gri);
SE = strel('kare', 3);
morfolojikGradient = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
maske = imbinarize(morfolojikGradient, 0.03);
SE = strel('kare', 2);
maske = kapat(maske, SE);
maske = dolgu(maske, 'delikler');
maske = bwareafilt(maske, 2); Alan gösterisinin % kontrol sayısı
notMask = ~maske;
maske = maske | bwpropfilt(notMask, 'Alan', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, maske, 'r'); % uygulama/işlev showMaskAsOverlay indirmeniz gerekiyor
BW2 = imfill(binaryImage, 'delikler');
new_image = BW2;
new_image(~maske) = 0; % arka planı ve delikleri ters çevir
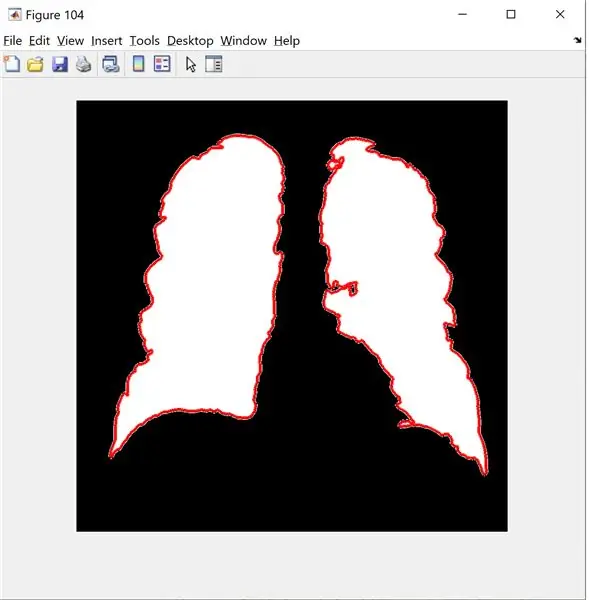
B=bwsınırları(yeni_görüntü); % sadece 2 boyutu kabul edebilir
şekil(104);
imshow(new_image);
devam etmek
sınır(B);
son
Adım 4: GUI Oluşturma
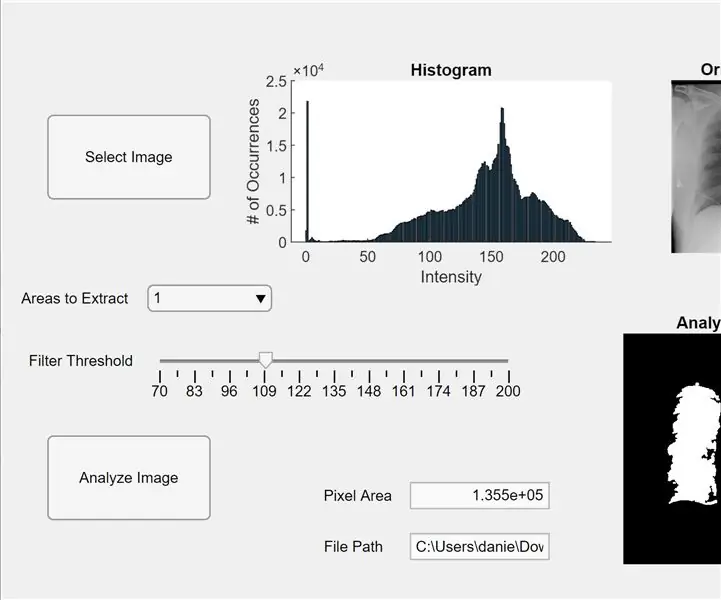
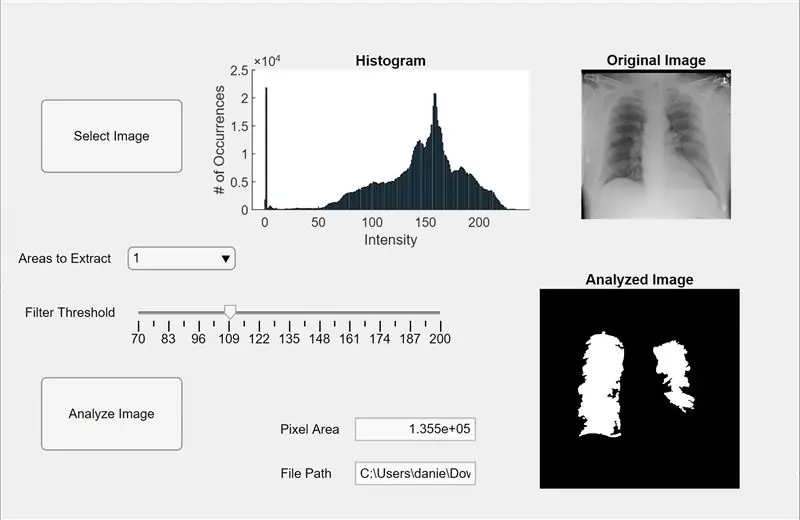
Şimdi, önceki kodu bir MATLAB uygulamasına entegre ediyoruz. MATLAB'de (Yeni > Uygulama) Uygulama Tasarımcısını açın. İlk olarak, ortadaki çalışma alanına üç eksende tıklayıp basılı tutup sürükleyerek arayüzü tasarlıyoruz. Ardından, iki düğme, bir düzenleme alanı (metin), bir düzenleme alanı (sayısal), bir kaydırıcı ve bir açılır menü, tıklayıp basılı tutup sürükleriz. İki eksenin her biri önizlemeyi gösterecek ve görüntüyü analiz edecek ve üçüncü eksenler, önizleme “seçili” görüntü için bir piksel histogramı gösterecektir. Düzenleme alanı (metin) kutusu, seçilen görüntünün dosya yolunu gösterecek ve düzenleme alanı (sayısal) akciğerlerin algılanan piksel alanını gösterecektir.
Şimdi App Designer'da tasarım görünümünden kod görünümüne geçin. Yanında artı işareti bulunan kırmızı “Özellikler” düğmesine tıklayarak özelliklerin kodunu koda girin. Aşağıda verilen kodda olduğu gibi I, eşik ve RegionToExtract özelliklerini başlatın. Ardından, çalışma alanının (Bileşen Tarayıcısı) sağ üst tarafındaki bir düğmeyi sağ tıklayın ve Geri Aramalar>Git… geri arama'dan gidin. “function SelectImageButtonPushed(app, event)” kodunu ekleyin. Bu kod, uigetfile kullanarak bilgisayarınızdan analiz etmek için bir görüntü seçmenize izin verir. Bir görüntü seçtikten sonra, bir histogram eşliğinde eksenlerin altında bir önizleme görüntüsü belirecektir. Ardından, diğer düğmeye sağ tıklayın ve bir geri arama işlevi oluşturmak için aynı prosedürü tekrarlayın.
"İşlev AnalyzeImageButtonPushed(app, event)" altındaki kodu ekleyin. Bu kod, görüntüyü analiz et düğmesi (bu kod için hangisini sağ tıkladıysanız) üzerine önizleme görüntüsünde piksel sayımı ve blob algılamayı gerçekleştirir. Düğmeleri programladıktan sonra şimdi kaydırıcıyı ve açılır menüyü programlayacağız. Kaydırıcıya sağ tıklayın, bir geri arama işlevi oluşturun ve sonuna kadar "function FilterThresholdSliderValueChanged(app, event)" altındaki kodu ekleyin. Bu, kaydırıcının gri yoğunluk eşiğini ayarlamasını sağlar.
Açılır menü için bir geri arama işlevi oluşturun ve açılır menünün analiz edilen görüntü eksenlerinde görüntülenen blob sayısını değiştirmesine izin vermek için "function AreastoExtractDropDownValueChanged(app, event)" altındaki kodu ekleyin. Şimdi, Bileşen Tarayıcısında her bir varlığa tıklayın ve varlık adlarını değiştirme, eksenleri kaldırma ve ölçeklemeyi değiştirme gibi özelliklerini beğeninize göre değiştirin. Tasarım Görünümünde Bileşen Tarayıcının varlıklarını işlevsel ve anlaşılması kolay bir düzene sürükleyip bırakın. Artık MATLAB'da akciğer görüntülerini piksel alanı için analiz edebilen bir uygulamanız var!
özellikler (Erişim = özel)I = ; % Görüntü dosyası
eşik = 257; Gri yoğunluğunu ikili hale getirmek için % eşiği
bölgelerToExtract = 2;
son
fonksiyon SelectImageButtonPushed(uygulama, olay)
clc;Dir = 'C:\Users\danie\Downloads\images_004\images'; %define değişmez dosyası "ön ek"
[imageExt, yol] = uigetfile('*.png'); % resim adının değişken kısmını al
imageName = [Dir filesep imageExt]; %concatenate değişmez ve değişken suşlar
app. I = imread(imageName); %resmi oku
imshow(app. I, 'ebeveyn', app. UIAxes); %görüntüyü göster
app. FilePathEditField. Value = yol; Orijinal görüntünün nereden geldiğini gösteren dosya yolunu %display
son
işlev AnalyzeImageButtonPushed(uygulama, olay)
orijinalResim = app. I;
orijinalResim = wiener2(app. I, [5 5]); %nokta kaldırma filtresi
histogram(app. AxesHistogram, app. I, 256); %görüntünün histogramını göster
a_thresh = orijinalImage >= app.threshold; % bu eşiği ayarla
labelImage = bwlabel(a_thresh);
sahne = bölgeprops(a_thresh, 'tümü');
sortedSolidity = sort([props. Solidity], 'iniş');
SB = sıralanmışSolidity(1);
SB == %1 ise SB yalnızca katılığı kabul eder ==1 kemikleri filtreler
SE = strel('kare', 3);
morfolojikGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
maske = imbinarize(morfolojikGradient, 0.03);
SE = strel('kare', 2);
maske = kapat(maske, SE);
maske = dolgu(maske, 'delikler');
maske = bwareafilt(maske, app.regionsToExtract);
Alan gösterisinin % kontrol sayısı
notMask = ~maske;
maske = maske | bwpropfilt(notMask, 'Alan', [-Inf, 5000 - eps(5000)]);
BW2 = imfill(labelImage, 'delikler');
new_image = BW2;
new_image(~maske) = 0;
B = bwsınırları(yeni_görüntü); % sadece 2 boyutu kabul edebilir imshow(new_image, 'parent', app. UIAxes2);
hold(app. UIAxes2, 'açık');
sınır(B);
set(gca, 'Ydir', 'ters');
akciğerArea = bwarea(new_image);
app. PixelAreaEditField. Value = akciğerArea;
son
son
function FilterThresholdSliderValueChanged(uygulama, olay)
app.threshold = app. FilterThresholdSlider. Value;
son
function AreastoExtractDropDownValueChanged(app, event)stringNumber = app. AreatoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
son
son
Önerilen:
MATLAB Kolay Yüz Algılama: 4 Adım

MATLAB Kolay Yüz Algılama: Bu talimatın temel amacı, görüntü işlemenin ne kadar kolay olacağını göstermektir, MATLABFace yardımıyla algılama ve izleme önemli ve aktif bir araştırma alanı olmuştur, bu yüzden açıklayacağım. nasıl yapılabilir ki
Matlab 2016b Nasıl Kodlanır ve Word'de Yayınlanır (Başlangıç Kılavuzu): 4 Adım

Matlab 2016b'yi Word'de Kodlama ve Yayınlama (Başlangıç Kılavuzu): Matlab, teknik sonuçları hesaplamak için kullanılan yüksek performanslı bir dil programıdır. Görselleri, hesaplamaları ve programlamayı kullanıcı dostu bir şekilde entegre etme yeteneğine sahiptir. Bu program ile kullanıcı sorunları ve çözümlerini yayınlayabilir
Raspberry Pi'yi Matlab Kullanarak Kontrol Etme: 5 Adım

Matlab Kullanarak Raspberry Pi'yi Kontrol Etme: Hey, bu eğitim matlab kullanarak başsız ahududu pi'nizi kontrol etmekle ilgilidir. Daha yeni ahududu pi kartını desteklemek için matlab'ı en son sürüme yüklemeniz gerekebilir
Matlab Tabanlı ROS Robotik Denetleyici: 9 Adım
Matlab tabanlı ROS Robotik Denetleyici: Çocukluğumdan beri hep Iron Man olmayı hayal ettim ve hala da öyle. Iron Man, insanlar bana gülse ya da bunun imkansız olduğunu söylese bile, gerçekçi olarak mümkün olan ve bir gün Iron Man olmayı arzuladığım karakterlerden biri
MATLAB'de Eğitim Programlama Geliştirmeleri: 6 Adım

MATLAB'de Tren Programlama Geliştirmeleri: Amaç: Bu programlanmış sistemin amacı, küçük ölçekli bir Arduino'ya bakmak ve Amtrak Demiryolu sistemlerinin belirli güvenlik özelliklerini potansiyel olarak geliştirmek için kodlamayı daha büyük ölçekte uygulamaktır. Bunu yapabilmek için toprak nemi sen
